Biox

Ein wichtiges Tool für Molecular Modeling-Anwendungen ist die Software Biox. Sie wurde von Max Dobler und dem Biografik Labor 3R der Universität Basel geschrieben und steht für Macintosh-, Linux- und Windowssysteme zur Verfügung. Ihre vielseitige Anwendung umfasst die Konstruktion, Analyse und Minimierung molekularer Strukturen. Im Rahmen des Molecular Modeling Kurses der FHNW wurde vor allem die Minimierungsfunktion rege benutzt. Diese Funktion berechnet Kraftfelder für Wasserstoffbrücken und Metall-Ligand-Wechselwirkungen. Mit Hilfe von Vorlagen für funktionelle Gruppen, Peptiden und Nukleinsäuren gestaltet sich das dreidimensionale Zeichnen von Strukturen einfach und schnell. Gezeichnete und minimierte Strukturen aus Biox sind mit anderen Tools wie Yetix und VirtualToxLab kompatibel.
- Quelle: http://biograf.ch/index.php?id=software&subid=bio, 11.09.2015
Yetix
Die Software Yetix wird für Computer-gestützte Verfahren in der modernen Arzneistoffentwicklung gebraucht. Das Computerprogramm wurde wie das Biox vom Biografiklabor 3R an der Universität in Basel unter der Leitung von Max Dobler und Angelo Vedani entwickelt. Die Software ist kompatibel mit allen Betriebssystemen sowie mit dem Biox und VirtualToxLab. Das Programm wird für interaktives 3D Docking verwendet und modelliert zusätzlich die Wechselwirkungen zwischen Proteinen (Rezeptor) und Molekülen (Target). Als Wechselwirkungen werden unter anderem Wasserstoffbrückenbindungen oder Metall-Ligand Interaktionen angezeigt. Für diesen Molecular Modeling Kurs wurde diese Software vor allem für Docking-Simulationen verwendet.
- Quelle: http://www.biograf.ch/index.php?id=software&subid=yeti, 11.09.2015
VirtualToxLab
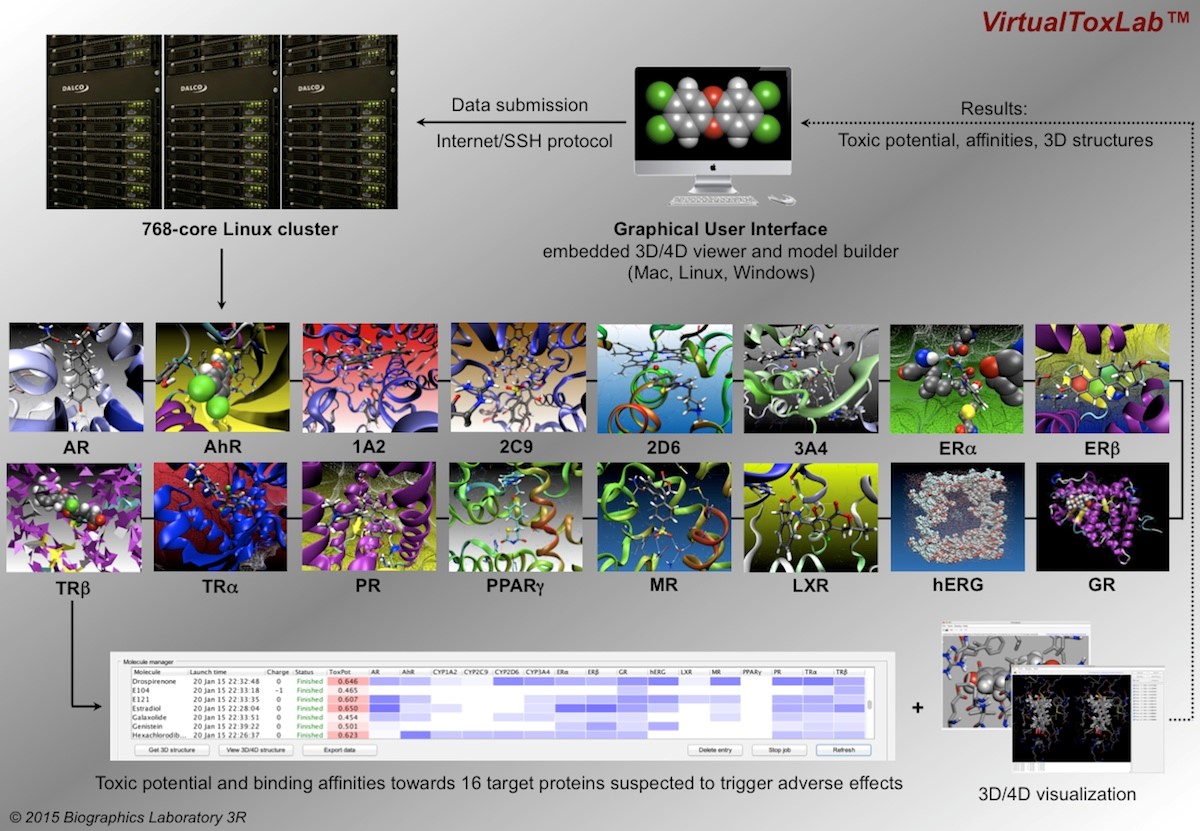
Erzeugung computerbasierter Voraussagen zum toxischen Potential von Chemikalien und Arzneistoffen. Mit der Computersoftware VirtualToxLab können potentiell toxische Stoffe ermittelt werden. Dank dieser Computersimulation können Tierversuche und teilweise sogar klinische Studien reduziert und/oder optimiert werden. Im besten Fall werden Tierversuche sogar überflüssig. Das Tool berechnet Wasserstoffbrückenbindungen und Bindungsaffinitäten von Liganden an 16 Rezeptoren. Diese Rezeptoren sind potentielle Bindungsstellen für endokrine und metabolische Störungen sowie Kanzerogenität und Kardiotoxizität. Die vom Programm vorausgesagte Bindung kann in Echtzeit in 3D oder 4D betrachtet werden. Daraus wird das toxische Potential berechnet, welches zwischen 0 und 1 liegt. Ein Wert unter 0.4 wird als unbedenklich bezeichnet. Liegt der Wert unter 0.6 sollte die Substanz sicherlich nicht über längere Zeit eingenommen werden und bei einem Wert über 0.6 muss mit akuten Gesundheitsfolgen gerechnet werden.
Im Kurs Molecular Modeling der FHNW an der Universität Basel diente die Software zur Bestimmung des toxischen Potentials und somit zur Eruierung von potentiell gefährlichen Wachstumsregulatoren. Die Software wurde unter der Leitung von Angelo Vedani, Max Dobler und Martin Smiesko im Biografik Labor 3R der Universität Basel entwickelt. Minimierte Strukturen aus BioX können einfach und praktisch ins VirtualToxLab eingelesen werden.
- Quelle: http://biograf.ch/index.php?id=projects&subid=virtualtoxlab, 11.09.2015
Consensus-Scoring
Für die Auswertung von physikalischen und toxischen Daten wurden die untenstehenden Programme bzw. Internetseiten verwendet.
- ToxTree: Toxtree ist ein einfaches Open Source Programm und wurde von Ideaconsult Ltd. entwickelt. Mithilfe der Molekülstruktur oder Eingabe der SMILES Nummer werden die Verbindungen in drei verschiedene Gefahrenkategorien zugeordnet: schwach (Klasse 1), mittel (Klasse 2) und stark (Klasse 3).
- Quelle: http://toxtree.sourceforge.net/, 11.09.2015
- Lazar: Diese Internetseite kann Voraussagen über Karzinogenität, Mutagenität, Letalität und Toxizität machen. Zum Ergebnis wird immer noch eine Zuverlässigkeit angegeben. Diese Seite basiert auf OpenTox und ist frei zugänglich.
- Quelle: http://lazar.in-silico.de/predict, 11.09.2015
- eADMET: Auf dieser Seite werden Aussagen über physikalisch-chemische Eigenschaften wie Löslichkeit, verschiedene Aktivität zu Enzym-Inhibition und logP-Wert gemacht.
- Quelle: http://www.eadmet.com/en/physprop.php, 11.09.2015
- Quick PK: Spezialisierte Applikation in der Schrodinger Suite für die Berechnung von relevanten Daten, wie molare Masse, logP, SASA usw.
- Quelle: http://131.152.93.108/cgi-bin/pkpredictor/pkpredict_2.py, 11.09.2015
- ChemID+: ChemID+ ist eine Internetseite, die das Marvin Scripts für die Berechnung von LogP, LogD bei pH 7.4, PSA, pKa und MWusw verwendet.
- Quelle: http://chem.sis.nlm.nih.gov/chemidplus/, 11.09.2015